本文由ChatGPT基于英文版翻译后稍加修改, 不准确之处以英文版为准.
在自然语言处理(NLP)研究的早期阶段,我们通过计算共享单词来量化文档之间的相似性。然而,这种简单的方法忽略了同义词它将“猫”和“小猫”视为毫无关联的实体,就像对待“猫”和“岩石”一样(图1,左侧)。由此产生的基于身份(identity)的单词表示,即独热(one-hot)编码,在应用于聚类文档时效果并不佳。幸运的是,随着Word2Vec等深度学习技术的出现 [1],NLP领域已经发生了变革。由于Word2Vec,曾经相距甚远的“猫”和“小猫”两个词现在被认为是几乎相同的(图1,右侧)。

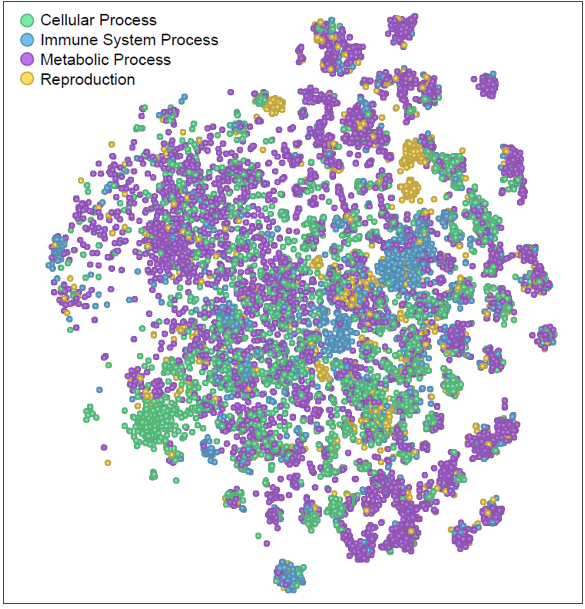
在基因分析中,我们一直面临着类似的困境。在比较基因列表时,我们计算共享的基因个数,类似于旧时的NLP方法。然而,像TLR7和MYD88这样在先天免疫信号中具有非常相似生物学作用的基因,当在出现在不同列表中时被当成是完全无关的而被忽视了。这就像“猫”和“小猫”曾经被认为是完全不同的一样。目前使用的基因列表的相似性算法很有局限。在我们最近的《自然通讯》发表的研究中 [2],我们将NLP与生物信息学进行了类比,首先我们引入了基因特征的功能性表示(Functional Representation of Gene Signatures — FRoGS),作为基因分析的Word2Vec等效方法(图2)。然后我们利用FRoGS的表征来揭示化合物的靶点。利用Broad的L1000数据集, 通过将shRNA/cDNA扰动的基因特征与中化合物的基因特征进行相似性比较,我们取得了显著的成果。具体而言,基于FRoGS的AI模型在识别真正的化合物靶点方面表现出了36%的召回率 (recall),超过了传统的独热编码方法所能达到的9%的召回率。
在这篇博客中,我们用两个浅显的例子进一步展示了FRoGS向量如何帮助实现强大的机器学习任务,将基因分析推向了传统独热编码不能够及的高度。
FRoGS能卓有成效地帮助机器学习的诀窍在哪里呢?因为基因的功能驱动着生物过程和它的表型。 ,它的功能被忽略了, AI模型就必须使出洪荒之力重新发现它们的功能 才能有效预测它们的表型— 而这个功能再发现的过程需要大量的训练数据。不幸的是,这样的大量数据并不总是可得到,于是有限的训练数据严重地影响了功能的学习从而影响了最终预测的准确性。FRoGS表征革命性地改变了基因的表示方式。每个人类基因都被编码为一个有意义的向量,既包含了在基因在Gene Ontology (GO [3])中注释的已知功能,也包含了从大规模转录组数据集(例如ARCHS4 [4])中蕴含的潜在功能 。FRoGS在AI模型的输入向量中注入功能信息, 模型无需浪费资源去从头开始学习基因的功能, 而可以专注于学习基因功能与表型之间复杂的关联。这样有限的训练数据全都被用在了刀刃上。
示例 1:组织特异性基因表达
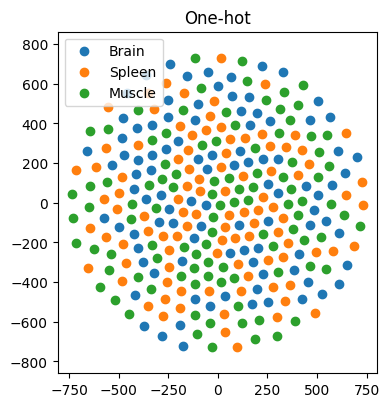
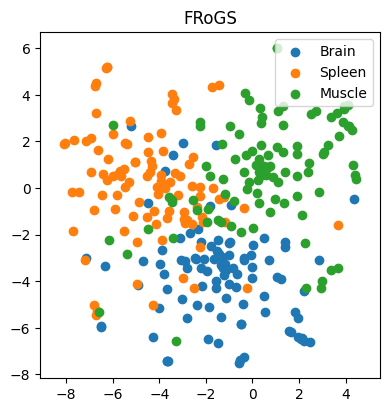
在我们的第一个示例中,我们分别使用97、98和100个在大脑、脾脏和肌肉中特异性表达的基因。我们的目标是预测基因表达的组织。传统上,独热编码将这些基因放置在高维空间中,基因之间没有任何的相似性。因此,AI模型难以学习有意义的模式,导致分类准确率仅约为29%(±3%)(n = 100次模拟)——类似于随机猜测(图3)。然而,当我们使用FRoGS向量表示基因时,t-SNE图中已经呈现了组织特异性聚类(图4),这样用机器学习找出分类的边界就很直接了。随机森林模型(Random Forest)的准确率飙升至约80%(±5%)(n = 100次模拟),展示了FRoGS使得AI模型即使在有限数据情况下也具有强大的学习能力(图4)。
示例 2:基因列表和功能特征
我们的数据集包括与动脉、心脏和大脑分别相关的35、24和122个基因列表。每个基因列表内含约100个基因。在独热编码方法中,基因列表被表示为其组成成员基因独热向量的总和。虽然这些基因列表也展示了一些聚类模式(图5),但与FRoGS基因特征嵌入形成的明显聚类相比,它们相形见绌(图6)。因此,用于预测的分类准确率为独热向量为85%(±4%)(n = 100次模拟),而FRoGS向量为100.0%(±0.4%)(n = 100次模拟)。


结论
FRoGS标志着生物信息学的一个新跳跃,类似于Word2Vec对NLP的影响。作为研究人员,让我们拥抱这一起探索FRoGS在不同生物问题中的应用潜力。请访问我们的GitHub代码库[5],在您的下一个机器学习尝试中发掘FRoGS的变革性能力。
引文